厚労省の新型コロナデータでグラフ作成
半年ぶりの投稿。
モジュールの使い方等いろいろ忘れているので調べながら思いだしつつ。
この投稿では、厚労省のサイトオープンデータ|厚生労働省
に置いてある新型コロナのデータセットcsvファイルをそのまま用いてグラフを描いてみます。
目次
実行環境
Androidスマホ
termux
Python3.8
JupyterNotebook
csvファイルはローカルにダウンロードして使用。
陽性者数
https://www.mhlw.go.jp/content/pcr_positive_daily.csv
# ライブラリのインポート import pandas as pd import numpy as np import matplotlib.pyplot as plt # Pandasでファイルの読み込み df_posi = pd.read_csv("pcr_positive_daily.csv")
# 上から5行だけデータフレームの確認
df_posi.head()
| 日付 | PCR 検査陽性者数(単日) | |
|---|---|---|
| 0 | 2020/1/16 | 1 |
| 1 | 2020/1/17 | 0 |
| 2 | 2020/1/18 | 0 |
| 3 | 2020/1/19 | 0 |
| 4 | 2020/1/20 | 0 |
# 欠損値の有無の確認
df_posi.isnull().sum()
日付 0
PCR 検査陽性者数(単日) 0
dtype: int64
# Numpy配列に変換 np_arr = np.array(df_posi) # 横・縦軸の配列データをx,yで x = np.array(np_arr[:,0]) #横軸データ y = np.array(np_arr[:,1]) #縦軸データ plt.plot(x,y) plt.savefig("pcr_positive_daily.png") plt.show()
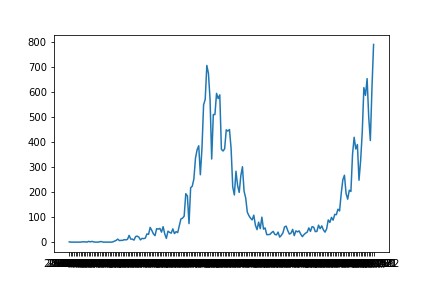
※横軸は見えにくいが「日付」です。
グラフは1日毎の新規感染者数を表している。政治家らが「第2波」と言っているのはこのグラフによるのだろう。
同様にして他のファイルもグラフ化していく。
PCR検査実施人数
https://www.mhlw.go.jp/content/pcr_tested_daily.csv
# Pandasデータフレームに読み込み df_tested = pd.read_csv("pcr_tested_daily.csv")
df_tested.head()
| 日付 | PCR 検査実施件数(単日) | |
|---|---|---|
| 0 | 2020/2/5 | 4 |
| 1 | 2020/2/6 | 19 |
| 2 | 2020/2/7 | 9 |
| 3 | 2020/2/8 | 4 |
| 4 | 2020/2/9 | 10 |
df_tested.isnull().sum()
日付 0
PCR 検査実施件数(単日) 0
dtype: int64
np_tested = np.array(df_tested) x_tested = np.array(np_tested[:,0]) y_tested = np.array(np_tested[:,1]) plt.plot(x_tested, y_tested) plt.savefig("pcr_tested_daily.png") plt.show()
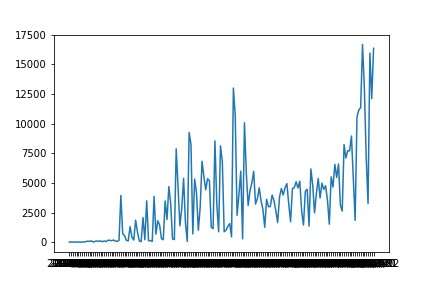
このグラフは1日毎のPCR検査実施人数を表している。
検査人数を増やしているので、前の感染者人数のグラフが増えたということです。小池都知事もそう発言している。
感染者の9割ほどは無症状と言われているが、検査人数を増やした為に無症状感染者が炙り出された形だと思われる。
初期段階から一定した人数で検査していた中で感染者の増加が再燃した、という訳ではない。
感染者は検査人数に比例して増えている。「第2波」は間違いだろう。
入院治療等を要する者の数
https://www.mhlw.go.jp/content/cases_total.csv
# データフレームに取り込み df_cases_total = pd.read_csv("cases_total.csv")
df_cases_total.head()
| 日付 | 入院治療を要する者 | |
|---|---|---|
| 0 | 2020/2/4 | 15 |
| 1 | 2020/2/5 | 16 |
| 2 | 2020/2/6 | 12 |
| 3 | 2020/2/7 | 12 |
| 4 | 2020/2/8 | 7 |
df_cases_total.isnull().sum()
日付 0
入院治療を要する者 0
dtype: int64
np_cases_total = np.array(df_cases_total) x_cases_total = np.array(np_cases_total[:,0]) y_cases_total =np.array(np_cases_total[:,1]) plt.plot(x_cases_total, y_cases_total) plt.savefig("cases_total.png") plt.show()

このグラフは入院治療を要すると判断された1日あたりの人数を表している。
検査人数を増やし、感染者がそれに比例して増加。それに伴い入院治療等の診断を下された人も増えたということ。
入院治療等の判断や症状の度合いについてはここでは不明。
退院または療養解除となった者の数
https://www.mhlw.go.jp/content/recovery_total.csv
# データフレームに取り込み df_recovery_total = pd.read_csv("recovery_total.csv")
df_recovery_total.head()
| 日付 | 退院、療養解除となった者 | |
|---|---|---|
| 0 | 2020/1/29 | 1 |
| 1 | 2020/1/30 | 1 |
| 2 | 2020/1/31 | 1 |
| 3 | 2020/2/1 | 1 |
| 4 | 2020/2/2 | 1 |
df_recovery_total.isnull().sum()
日付 0
退院、療養解除となった者 0
dtype: int64
np_recovery_total = np.array(df_recovery_total) x_recovery_total = np.array(np_recovery_total[:,0]) y_recovery_total = np.array(np_recovery_total[:,1]) plt.plot(x_recovery_total, y_recovery_total) plt.savefig("recovery_total.png") plt.show()
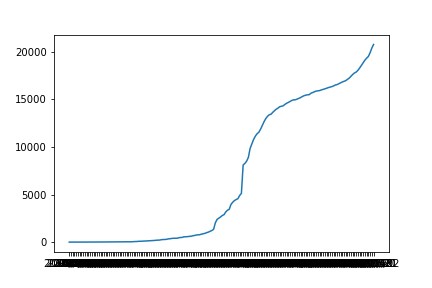
このグラフは、退院と療養解除の人数を積算で表している。
死亡者数
https://www.mhlw.go.jp/content/death_total.csv
# データフレームに取り込み df_death_total = pd.read_csv("death_total.csv")
df_death_total
| 日付 | 死亡者数 | |
|---|---|---|
| 0 | 2020/2/14 | 1 |
| 1 | 2020/2/15 | 1 |
| 2 | 2020/2/16 | 1 |
| 3 | 2020/2/17 | 1 |
| 4 | 2020/2/18 | 1 |
| ... | ... | ... |
| 155 | 2020/7/18 | 984 |
| 156 | 2020/7/19 | 984 |
| 157 | 2020/7/20 | 987 |
| 158 | 2020/7/21 | 988 |
| 159 | 2020/7/22 | 989 |
160 rows × 2 columns
# Numpy配列に変換
np_death_total = np.array(df_death_total)
# 死者数の積算の列を抜き出し np_y_total = np_death_total[:,1]
# 累積を差分にする(1日毎の増加分に変換)
np_diff = np.diff(np_y_total)
np_diff
array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 2, 1, 1, 0, 1, 0, 0, 0, 0, 0,
0, 1, 2, 3, 3, 4, 2, 1, 2, 4, 1, 2, 2, 2, 1, 5, 1, 1, 2, 1, 3, 3,
2, 2, 1, 3, 3, 6, 1, 3, 7, 1, 4, 3, 6, 4, 4, 7, 10, 17, 12, 6, 7,
10, 15, 91, 10, 30, 17, 14, 3, 25, 13, 26, 17, 26, 34, 18, 11, 22,
8, 49, 13, 8, 22, 25, 19, 23, 15, 19, 5, 14, 8, 6, 19, 12, 12, 10,
16, 12, 9, 7, 12, 5, 1, 2, 6, 3, 4, 7, 2, 0, 0, 3, 1, 2, 2, 1, 0,
2, 4, 4, 0, 17, 1, 0, 2, 8, 4, 1, 2, 0, 1, 0, 2, 1, 1, 1, 0, 0, 1,
2, 1, 1, 0, 0, 0, 0, 2, 1, 0, 0, 0, 0, 3, 1, 1], dtype=object)
# 最大値の確認
np_diff.max()
91
# 先頭に初期値を追加 y_death_daily = np.append(1,np_diff) x_death_daily = np.array(np_death_total[:,0]) plt.plot(x_death_daily, y_death_daily) plt.savefig("death_daily.png") plt.show()
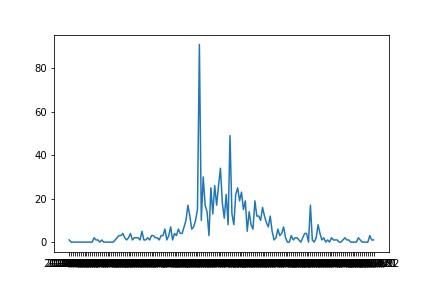
このグラフは新型コロナが原因と判断された死亡者の1日毎の人数を表している。
基データcsvファイルは死亡者数の積算になっているため、np.diffを使って1日あたりの数に変換した。データが一つ減るので、先頭にnp.appendで初期値の1(1人)を追加している。
前のグラフに於けるいわゆる「第2波」部分で検査人数や感染者数が増えているにもかかわらず、死者数の増加はこのグラフでは見られない。
基本的に死亡原因の診断は各医師の判断に任されているようです。
ウイルス(コロナなど)が原因の風邪をこじらせて肺炎を併発して亡くなった場合、死亡診断は「肺炎」とされる可能性が高い。そうであるから肺炎の死亡者数が年間10万人以上いると言える。
「風邪は万病の基」ということで。
PCR検査の実施件数
https://www.mhlw.go.jp/content/pcr_case_daily.csv
df_pcr_case_daily = pd.read_csv("pcr_case_daily.csv")
df_pcr_case_daily.head()
| 日付 | 国立感染症研究所 | 検疫所 | 地方衛生研究所・保健所 | 民間検査会社 | 大学等 | 医療機関 | |
|---|---|---|---|---|---|---|---|
| 0 | 2020/2/18 | 472 | 75 | 398 | 0 | 79 | NaN |
| 1 | 2020/2/19 | 15 | 68 | 609 | 0 | 0 | NaN |
| 2 | 2020/2/20 | 20 | 15 | 758 | 0 | 0 | NaN |
| 3 | 2020/2/21 | 261 | 188 | 902 | 132 | 108 | NaN |
| 4 | 2020/2/22 | 341 | 127 | 677 | 2 | 19 | NaN |
df_pcr_case_daily.isnull().sum()
日付 0
国立感染症研究所 0
検疫所 0
地方衛生研究所・保健所 0
民間検査会社 0
大学等 0
医療機関 17
dtype: int64
どこでどれだけ検査の実施がされたかのデータ。グラフは無しで。
発生状況
https://www.mhlw.go.jp/content/current_situation.csv
df_current_situation = pd.read_csv("current_situation.csv")
df_current_situation.head()
| Unnamed: 0 | PCR検査\n実施人数 ※3 | 陽性者数 | 入院治療等を要する者の数 | うち重症者の数 | 退院又は療養解除と\nなった者の数 | 死亡者数 | 確認中 ※4 | |
|---|---|---|---|---|---|---|---|---|
| 0 | 国内事例 ※1,※5\n(チャーター便帰国\n者を除く) | 579,185\n(+16,357) | 26,529\n(+792) ※2 | 4,751\n(+330) | 59\n(+4) | 20,787\n(+384) | 989\n(+1) | 14\n(+7) |
| 1 | 空港検疫 | 108,022\n(+1,060) | 485\n(+4) | 251\n(+4) | 0 | 233 | 1 | 0 |
| 2 | チャーター便\n帰国者事例 | 829 | 15 | 0 | 0 | 15 | 0 | 0 |
| 3 | 合計 | 688,036\n(+17,417) | 27,029\n(+796) ※2 | 5,002\n(+334) | 59\n(+4) | 21,035\n(+384) | 990\n(+1) | 14\n(+7) |
これもグラフ無しで。
以上。