厚労省新型コロナ7月27日迄のデータによるグラフ作成
厚労省のサイト オープンデータ|厚生労働省 に掲載の新型コロナのデータCSVファイルからグラフを作成します。
7/28更新で27日までのデータが配信されております。
前回は7/22までのデータ
厚労省の新型コロナデータでグラフ作成 - よちよちpython
目次
実行環境
Androidスマホ
termux
Python3.8
JupyterNotebook
新型コロナのデータCSVファイルは上記の厚労省オープンデータよりローカルにダウンロードして使用。
陽性者数
https://www.mhlw.go.jp/content/pcr_positive_daily.csv
# ライブラリのインポート import pandas as pd import numpy as np import matplotlib.pyplot as plt
# ファイルパス filename = "pcr_positive_daily.csv" # ファイル読み込みデータフレームに df_posi = pd.read_csv(filename) # 出力 df_posi
| 日付 | PCR 検査陽性者数(単日) | |
|---|---|---|
| 0 | 2020/1/16 | 1 |
| 1 | 2020/1/17 | 0 |
| 2 | 2020/1/18 | 0 |
| 3 | 2020/1/19 | 0 |
| 4 | 2020/1/20 | 0 |
| ... | ... | ... |
| 189 | 2020/7/23 | 966 |
| 190 | 2020/7/24 | 766 |
| 191 | 2020/7/25 | 798 |
| 192 | 2020/7/26 | 830 |
| 193 | 2020/7/27 | 581 |
194 rows × 2 columns
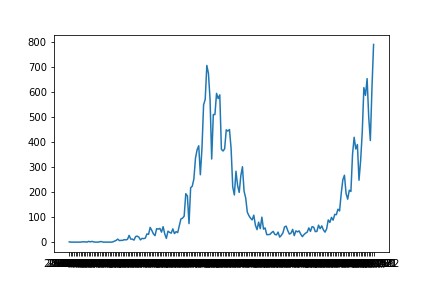
2020/1/16~2020/7/27迄の1日毎のPCR検査陽性者数のデータが入っている。
# データフレームをNumpy配列に変換 np_posi = np.array(df_posi) # 横軸と縦軸の配列に分ける x_posi = np.array(np_posi[:,0]) #横軸(日付) y_posi = np.array(np_posi[:,1]) #縦軸(陽性者数) # グラフ作図 plt.plot(x_posi, y_posi) plt.xticks(x_posi[::16], rotation=45) #横軸の表示変更 plt.savefig("pcr_positive_daily.png") # グラフの保存 plt.show() # グラフをJupyterNotebookに表示

このグラフは、1日毎のPCR検査の新規陽性者数を表している。
plt.xticks(x_posi[::16], rotation=45)の部分では、横軸の表示を減らす為に16日間隔(適当)にし、日付も45度回転させて見やすくした。
この部分を書かないと、前回 厚労省の新型コロナデータでグラフ作成 - よちよちpython の↓のグラフのように、日付が重なり黒く塗りつぶれてしまう。
以下、同様にして他のデータもグラフを作成していく。
PCR検査実施人数
https://www.mhlw.go.jp/content/pcr_tested_daily.csv
# Pandasデータフレームに読み込み df_tested = pd.read_csv("pcr_tested_daily.csv") # データフレームの表示 df_tested
| 日付 | PCR 検査実施件数(単日) | |
|---|---|---|
| 0 | 2020/2/5 | 4 |
| 1 | 2020/2/6 | 19 |
| 2 | 2020/2/7 | 9 |
| 3 | 2020/2/8 | 4 |
| 4 | 2020/2/9 | 10 |
| ... | ... | ... |
| 166 | 2020/7/23 | 8310 |
| 167 | 2020/7/24 | 5695 |
| 168 | 2020/7/25 | 9530 |
| 169 | 2020/7/26 | 3785 |
| 170 | 2020/7/27 | 24182 |
171 rows × 2 columns
2020/2/5~7/27迄の1日毎のPCR検査実施件数のデータが入っている。
# Numpy配列に変換 np_tested = np.array(df_tested) # 横軸と縦軸の配列に分ける x_tested = np.array(np_tested[:,0]) y_tested = np.array(np_tested[:,1]) # グラフ作図 plt.plot(x_tested, y_tested) plt.xticks(x_tested[::14],rotation=45) plt.savefig("pcr_tested_daily.png") plt.show()

このグラフは1日毎のPCR検査実施件数を表している。
だいぶん増やしている。それに比例して前のグラフでの陽性者数も伸びている。
この調子で今後も陽性者数が増えていくだろう。
ギザギザしてるのは消毒する為に間を空かしてるとかかな?
入院治療等を要する者の数
https://www.mhlw.go.jp/content/cases_total.csv
# データフレームに取り込み df_cases_total = pd.read_csv("cases_total.csv") # データフレームの表示 df_cases_total
| 日付 | 入院治療を要する者 | |
|---|---|---|
| 0 | 2020/2/4 | 15 |
| 1 | 2020/2/5 | 16 |
| 2 | 2020/2/6 | 12 |
| 3 | 2020/2/7 | 12 |
| 4 | 2020/2/8 | 7 |
| ... | ... | ... |
| 170 | 2020/7/23 | 5369 |
| 171 | 2020/7/24 | 5949 |
| 172 | 2020/7/25 | 6315 |
| 173 | 2020/7/26 | 6573 |
| 174 | 2020/7/27 | 6828 |
175 rows × 2 columns
2020/2/4~7/27迄の1日あたり入院治療等を要する者の数のデータ。
# Numpy配列に変換 np_cases_total = np.array(df_cases_total) # 横軸と縦軸の配列に分ける x_cases_total = np.array(np_cases_total[:,0]) y_cases_total = np.array(np_cases_total[:,1]) # グラフ作図 plt.plot(x_cases_total, y_cases_total) plt.xticks(x_cases_total[::14],rotation=45) plt.savefig("cases_total.png") plt.show()

このグラフは1日あたりの入院治療等を要する者の数を表している。
- 5/7以前は入院者数を、
- それ以降は入院治療等を要する者
と内容が変わっていると オープンデータ|厚生労働省 に説明がある。
5/8以降の数字は実際に入院している人数ではない。軽度の若者などをいちいち入院させていると直ぐに病院のキャパシティをオーバーしてしまうね。
退院または治療解除となった者の数
https://www.mhlw.go.jp/content/recovery_total.csv
# データフレームに取り込み df_recovery = pd.read_csv("recovery_total.csv") # データフレーム表示 df_recovery
| 日付 | 退院、療養解除となった者 | |
|---|---|---|
| 0 | 2020/1/29 | 1 |
| 1 | 2020/1/30 | 1 |
| 2 | 2020/1/31 | 1 |
| 3 | 2020/2/1 | 1 |
| 4 | 2020/2/2 | 1 |
| ... | ... | ... |
| 176 | 2020/7/23 | 21080 |
| 177 | 2020/7/24 | 21319 |
| 178 | 2020/7/25 | 21514 |
| 179 | 2020/7/26 | 21868 |
| 180 | 2020/7/27 | 22560 |
181 rows × 2 columns
2020/1/29~7/27迄の退院または治療解除となった者の積算データが入っている。
# Numpy配列に変換 np_recovery = np.array(df_recovery) # 横軸と縦軸の配列に分ける x_recovery = np.array(np_recovery[:,0]) y_recovery = np.array(np_recovery[:,1]) # グラフ作図 plt.plot(x_recovery, y_recovery) plt.xticks(x_recovery[::14], rotation=45) plt.savefig("recovery_total.png") plt.show()

このグラフは退院または治療解除となった者の数を積算で表している。
死亡者数
https://www.mhlw.go.jp/content/death_total.csv
積算の死亡者数
# データフレームに取り込み df_death = pd.read_csv("death_total.csv") # 表示 df_death
| 日付 | 死亡者数 | |
|---|---|---|
| 0 | 2020/2/14 | 1 |
| 1 | 2020/2/15 | 1 |
| 2 | 2020/2/16 | 1 |
| 3 | 2020/2/17 | 1 |
| 4 | 2020/2/18 | 1 |
| ... | ... | ... |
| 160 | 2020/7/23 | 991 |
| 161 | 2020/7/24 | 992 |
| 162 | 2020/7/25 | 995 |
| 163 | 2020/7/26 | 995 |
| 164 | 2020/7/27 | 997 |
165 rows × 2 columns
2/14~7/27の新型コロナによる死亡者数の積算データ。
# Numpy配列に変換 np_death = np.array(df_death) # 横軸と縦軸の配列に分ける x_death = np.array(np_death[:,0]) y_death = np.array(np_death[:,1]) # グラフ作図 plt.plot(x_death, y_death) plt.xticks(x_death[::14], rotation=45) plt.savefig("death_total.png") plt.show()

このグラフは、2/14~7/27の新型コロナによる死亡者数の積算を表している。6月あたりから横ばい。
1日毎の死亡者数のグラフも描いてみます。
1日毎の死亡者数
# データフレームのNumpy配列 # np_death = np.array(df_death) # 横軸の配列 x_death = np.array(np_death[:,0]) # 縦軸は1日毎の配列に変換 y_death_daily = np.append(1, np.diff(np_death[:,1])) # 1日毎の死亡者数の配列 y_death_daily
array([1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 2, 1, 1, 0, 1, 0, 0, 0, 0,
0, 0, 1, 2, 3, 3, 4, 2, 1, 2, 4, 1, 2, 2, 2, 1, 5, 1, 1, 2, 1, 3,
3, 2, 2, 1, 3, 3, 6, 1, 3, 7, 1, 4, 3, 6, 4, 4, 7, 10, 17, 12, 6,
7, 10, 15, 91, 10, 30, 17, 14, 3, 25, 13, 26, 17, 26, 34, 18, 11,
22, 8, 49, 13, 8, 22, 25, 19, 23, 15, 19, 5, 14, 8, 6, 19, 12, 12,
10, 16, 12, 9, 7, 12, 5, 1, 2, 6, 3, 4, 7, 2, 0, 0, 3, 1, 2, 2, 1,
0, 2, 4, 4, 0, 17, 1, 0, 2, 8, 4, 1, 2, 0, 1, 0, 2, 1, 1, 1, 0, 0,
1, 2, 1, 1, 0, 0, 0, 0, 2, 1, 0, 0, 0, 0, 3, 1, 1, 2, 1, 3, 0, 2],
dtype=object)
# グラフ作図 plt.plot(x_death, y_death_daily) plt.xticks(x_death[::14], rotation=45) plt.savefig("death_daily.png") plt.show()

このグラフは、1日毎の新型コロナ死亡者数を表している。
基データが積算だった為、y_death_daily = np.append(1, np.diff(np_death[:,1]))
の部分で1日毎の死亡者数に変換した。
np.diff(配列) で差分を出す。これで1日毎の増加分が出せる。データがひとつ減るので、np.append(追加データ, 元の配列 )で初日のデータを追加している。元の配列の先頭にデータ(2/14の1人)が追加される。
例) np.diff(配列)の使い方
# 積算っぽい配列を適当に作る np_sekisan = np.array([1,5,8,9,14]) # 積算の配列を表示 print("積算", np_sekisan) # 差分を出す np_sabun = np.diff(np_sekisan) #差分の配列を表示 print("差分", np_sabun) # 増加分の配列に変換 np_zouka = np.append(1, np_sabun) # 増加分の配列を表示 print("増加分", np_zouka)
積算 [ 1 5 8 9 14]
差分 [4 3 1 5]
増加分 [1 4 3 1 5]
PCR検査の実施件数
https://www.mhlw.go.jp/content/pcr_case_daily.csv
# データフレームに取り込み df_pcr_case_daily = pd.read_csv("pcr_case_daily.csv") # 表示 df_pcr_case_daily
| 日付 | 国立感染症研究所 | 検疫所 | 地方衛生研究所・保健所 | 民間検査会社 | 大学等 | 医療機関 | |
|---|---|---|---|---|---|---|---|
| 0 | 2020/2/18 | 472 | 75 | 398 | 0 | 79 | NaN |
| 1 | 2020/2/19 | 15 | 68 | 609 | 0 | 0 | NaN |
| 2 | 2020/2/20 | 20 | 15 | 758 | 0 | 0 | NaN |
| 3 | 2020/2/21 | 261 | 188 | 902 | 132 | 108 | NaN |
| 4 | 2020/2/22 | 341 | 127 | 677 | 2 | 19 | NaN |
| ... | ... | ... | ... | ... | ... | ... | ... |
| 155 | 2020/7/22 | 0 | 1066 | 3123 | 12909 | 1676 | 1263.0 |
| 156 | 2020/7/23 | 121 | 1083 | 2963 | 4245 | 361 | 589.0 |
| 157 | 2020/7/24 | 0 | 1237 | 2648 | 1984 | 165 | 607.0 |
| 158 | 2020/7/25 | 0 | 1331 | 2190 | 7824 | 691 | 966.0 |
| 159 | 2020/7/26 | 0 | 1492 | 2695 | 3101 | 172 | 448.0 |
160 rows × 7 columns
2/18~7/26までで各医療機関での1日毎の検査実施件数。グラフは省略。
発生状況
https://www.mhlw.go.jp/content/current_situation.csv
# データフレームに取り込み df_situation = pd.read_csv("current_situation.csv") # 表示 df_situation
| Unnamed: 0 | PCR検査\n実施人数 ※3 | 陽性者数 | 入院治療等を要する者の数 | うち重症者の数 | 退院又は療養解除と\nなった者の数 | 死亡者数 | 確認中 ※4 | |
|---|---|---|---|---|---|---|---|---|
| 0 | 国内事例 ※1,※5\n(チャーター便帰国\n者を除く) | 630,687\n(24,182) | 30,406\n(+581) ※2 | 6,828\n(+255) | 76\n(+9) | 22,560\n(+692) | 997\n(+2) | 27\n(+5) |
| 1 | 空港検疫 | 114,713\n(+1,546) | 540\n(+18) | 303\n(+15) | 0 | 236\n(+3) | 1 | 0 |
| 2 | チャーター便\n帰国者事例 | 829 | 15 | 0 | 0 | 15 | 0 | 0 |
| 3 | 合計 | 746,229\n(+25,728) | 30,961\n(+599) ※2 | 7,131\n(+270) | 76\n(+9) | 22,811\n(+695) | 998\n(+2) | 27\n(+5) |
データをまとめたものらしい。
グラフは省略。重病者は76(+9)名とある。
おわりに
昨年の熱中症による死亡者数は1500人ほど。インフルエンザは毎年患者数が1000万人以上出ていて、死亡者数も3000人~多い年では数万人が亡くなる。肺炎による死亡者は年間10万人以上。
それらと比較すると、コロナはなんとも非力で、騒動のやり過ぎ感が激しい。
メディアは初期段階から感染者数とレアケースばかり報道して恐怖を巻き散らして来た。
それによって、全国の企業や個人事業者を数百社つぶし、50兆円ほど吹き飛ばし、リーマンショックの80倍の生活困窮者を出している。最新設備の整った病院が全国に幾つも建てられる金額よ。
コロナ自体より過剰な騒動の方が遥かに甚大な被害を出している。明らかに政治家とマスコミと学者と自粛工作員による人災。と思いますけど。