【Pandas resample()】月別集計、時系列データを期間ごとに集計する方法
「売上データを月別で集計したい!」
というケースはよくありますよね。
今回は、Pandasを使って時系列データを週や月や四半期など期間ごとに集計する方法をやります。
実験室のセンサーや天候観測、売上やコロナのデータのように、秒単位や日毎で何らかのデータが連続的に入ったものを時系列データなどと呼びます。これらを年、月、週、四半期の期間ごとで合計を出したいような場面は頻繁にあるかと思います。
Pandasでデータファイルを読み込み、resample()メソッドを使うと簡単にできます。
なかなか便利です。覚えておくと重宝しそうですよ!
目次
- 目次
- 実行環境
- 手順
- 使用するデータ
- 1. pd.read_csv()でネット上からファイルを読み込む
- 2. 読み込んだファイルをローカルに保存
- 3. ローカルに保存したファイルを読み込む
- 4. resample()メソッドを使った期間の集計
- 5. 月ごと集計したグラフの作成
- おまけ 陽性率を算出
実行環境
- Windows10 WSLのUbuntu上のAnaconda
- Jupyter Notebook
- Python3.8
- 外部ライブラリ
- Pandas
- Matplotlib
- Pandas
今更だが、WSLでUbuntu Linuxの文字フォントがLinux臭い…
(Windows側に入れてあるVSCodeのリモートのは通常通り)
手順
今回行う手順は以下の通りです。
- Pandasのpd.read_csv()でCSVファイルをネット上から直接読み込みます。
- 一応、df.to_csv()でファイルをローカルに保存します。
- ローカルに保存したファイルを読み込む。
- df.resample().sum()で期間ごとの集計を出します。
- グラフを作成します。
使用するデータ
厚生労働省のコロナのサイトオープンデータ|厚生労働省 と東洋経済オンラインから。
今回は日毎の陽性者数と検査実施件数だけを使いますが、年代別のコロナデータのCSVファイルなども載せておきます。
# コロナのデータCSVファイル 厚労省/東洋経済より URLs = ["https://www.mhlw.go.jp/content/pcr_positive_daily.csv", # ⓪ PCR陽性者数 厚労省 "https://www.mhlw.go.jp/content/pcr_tested_daily.csv", # ① PCR検査実施人数 厚労省 "https://www.mhlw.go.jp/content/cases_total.csv", # ② 入院治療等を要する者の数 厚労省 "https://www.mhlw.go.jp/content/recovery_total.csv", # ③ 退院又は治療解除となった者の数 厚労省 "https://www.mhlw.go.jp/content/death_total.csv", # ④ 死亡者数合計 厚労省 "https://www.mhlw.go.jp/content/pcr_case_daily.csv", # ⑤ PCR検査の実施件数 厚労省 "https://www.mhlw.go.jp/content/current_situation.csv", # ⑥ 発生状況 厚労省 "https://www.mhlw.go.jp/content/severe_daily.csv", # ⑦ 重傷者数 厚労省 "https://toyokeizai.net/sp/visual/tko/covid19/csv/demography.csv"] # ⑧ 年齢別合計(陽性者、入院者、重症者、死亡者) 東洋経済 # CSVファイル名抽出用関数 辞書型 def make_savenames(URLs): savenames = {} for i, fname in enumerate(URLs): savenames[i] = fname.split('/')[-1] return savenames print(make_savenames(URLs))
{0: 'pcr_positive_daily.csv', 1: 'pcr_tested_daily.csv', 2: 'cases_total.csv', 3: 'recovery_total.csv', 4: 'death_total.csv', 5: 'pcr_case_daily.csv', 6: 'current_situation.csv', 7: 'severe_daily.csv', 8: 'demography.csv'}
あとで使うかなと思って関数を作っておいた。
1. pd.read_csv()でネット上からファイルを読み込む
上に掲載したCSVファイルの⓪番目のPCR陽性者数と、①番目のPCR検査実施件数をPandasで読み込みます。
import pandas as pd # PCR陽性者数ファイルの読込 df_positive = pd.read_csv(URLs[0]) # PCR検査実施件数ファイルの読込 df_tested = pd.read_csv(URLs[1])
2. 読み込んだファイルをローカルに保存
2.1 読み込んだファイルをローカルに保存
何度も使うかもしれませんので、ファイルを一旦ローカルに保存したいと思います。
pd.DataFrame.to_csv(保存名)で保存します。このまま保存すると読み込んだ際に自動的に振られた0番から始まるindexが左端の列に書き込まれてしまうので、引数でindex=Falseを指定します。保存名は上で用意したURLからファイル名を取り出す関数を使います。
# 保存名 savenames = make_savenames(URLs) # PCR陽性者数ファイルを保存 df_positive.to_csv(savenames[0], index=False) # PCR検査実施件数ファイルを保存 df_tested.to_csv(savenames[1], index=False)
これで保存できました。
3. ローカルに保存したファイルを読み込む
保存せずに直接ネットから読み込む場合と方法は同じです。
ただ、この後みていくように、pd.read_csv()の引数をいろいろ変えると読み込まれたデータフレームの表示がどのように変わるかを調べたりする時に毎回ネットからダウンロードするのはアレなので、ローカル保存しました。
# PCR陽性者数ファイル読込 df_posi = pd.read_csv(savenames[0]) df_posi
| 日付 | PCR 検査陽性者数(単日) | |
|---|---|---|
| 0 | 2020/1/16 | 1 |
| 1 | 2020/1/17 | 0 |
| 2 | 2020/1/18 | 0 |
| 3 | 2020/1/19 | 0 |
| 4 | 2020/1/20 | 0 |
| ... | ... | ... |
| 483 | 2021/5/13 | 6867 |
| 484 | 2021/5/14 | 6269 |
| 485 | 2021/5/15 | 6420 |
| 486 | 2021/5/16 | 5247 |
| 487 | 2021/5/17 | 3677 |
488 rows × 2 columns
普通に読み込むと(引数無しで読むと)インデックス番号が0から割り振られ、自動的に左端に付いています。
◆型を見てみます。
# 型確認
df_posi.dtypes
日付 object
PCR 検査陽性者数(単日) int64
dtype: object
日付はobject型になっています。これがdatetime型でないと期間ごとの集計が上手く行きません。これはあとで見ていきます
3.1 日付をdatetime型で、かつインデックスとして読み込むには
インデックスを0番から自動で割り振られる連番ではなく、データに入っている日付列に変更します。
◆方法としては、
- pd.read_csv()でファイルを読み込む場合に、
- 引数で
index_col=インデックス列番号とparse_dates=Trueを指定する
- 引数で
- pd.read_csv()で引数を指定せずに読み込んだ場合は、
- dfの日付の型を
pd.to_datetime(df)でdatetime型に直し、 set_index()で日付列をインデックスにセットする
- dfの日付の型を
3.1.1 まず、1の引数に指定して読み込む方法
# 0列目(日付)をインデックスにし、日付をdatetime型に変換して読み込む df_posi2 = pd.read_csv(savenames[0], index_col=0, parse_dates=True) df_posi2
| PCR 検査陽性者数(単日) | |
|---|---|
| 日付 | |
| 2020-01-16 | 1 |
| 2020-01-17 | 0 |
| 2020-01-18 | 0 |
| 2020-01-19 | 0 |
| 2020-01-20 | 0 |
| ... | ... |
| 2021-05-13 | 6867 |
| 2021-05-14 | 6269 |
| 2021-05-15 | 6420 |
| 2021-05-16 | 5247 |
| 2021-05-17 | 3677 |
488 rows × 1 columns
最初に引数無しで読み込んだデータフレーム(df_posi)の表示とは幾つかの点で変わっています。
- 日付がインデックスになった。
- 日付表示が「2020/1/16」→「2020-01-16」になった。
◆型の確認をしておきます。
# 型の確認
df_posi2.dtypes
PCR 検査陽性者数(単日) int64
dtype: object
これでdatetime型のインデックスに変換して読み込むことができました。
3.1.2 先にpd.read_csv()で読み込んでしまった場合
引数無指定で普通に読み込んだ後、インデックスとdatetime型への変換です。
# おっと、引数を指定せず、普通に読み込んでしまった! df_posi_r = pd.read_csv(savenames[0]) df_posi_r
| 日付 | PCR 検査陽性者数(単日) | |
|---|---|---|
| 0 | 2020/1/16 | 1 |
| 1 | 2020/1/17 | 0 |
| 2 | 2020/1/18 | 0 |
| 3 | 2020/1/19 | 0 |
| 4 | 2020/1/20 | 0 |
| ... | ... | ... |
| 483 | 2021/5/13 | 6867 |
| 484 | 2021/5/14 | 6269 |
| 485 | 2021/5/15 | 6420 |
| 486 | 2021/5/16 | 5247 |
| 487 | 2021/5/17 | 3677 |
488 rows × 2 columns
引数無しで読み込んでしまい、0から始まる連番のインデックスがついたデータフレームができた。
◆日付のインデックスにするには以下のように行います。
# 日付列をdatetimeに変換 pd.to_datetime(df_posi_r['日付'])
0 2020-01-16
1 2020-01-17
2 2020-01-18
3 2020-01-19
4 2020-01-20
...
483 2021-05-13
484 2021-05-14
485 2021-05-15
486 2021-05-16
487 2021-05-17
Name: 日付, Length: 488, dtype: datetime64[ns]
# 日付をdatetime型に変換し、インデックスにする df_posi_r['日付'] = pd.to_datetime(df_posi_r['日付']) df_posi_r = df_posi_r.set_index(df_posi_r['日付']) # 表示 df_posi_r
| 日付 | PCR 検査陽性者数(単日) | |
|---|---|---|
| 日付 | ||
| 2020-01-16 | 2020-01-16 | 1 |
| 2020-01-17 | 2020-01-17 | 0 |
| 2020-01-18 | 2020-01-18 | 0 |
| 2020-01-19 | 2020-01-19 | 0 |
| 2020-01-20 | 2020-01-20 | 0 |
| ... | ... | ... |
| 2021-05-13 | 2021-05-13 | 6867 |
| 2021-05-14 | 2021-05-14 | 6269 |
| 2021-05-15 | 2021-05-15 | 6420 |
| 2021-05-16 | 2021-05-16 | 5247 |
| 2021-05-17 | 2021-05-17 | 3677 |
488 rows × 2 columns
1番目と2番目の方法でデータフレームの表示が異なっています。2番目のは日付列が付いたまま。
◆列を削除したい場合は次のようにすればおk。
# 念のためコピー df_posi3 = df_posi2.copy() # dropで日付列を削除 df_posi3.drop(df_posi3.index[[0]], axis=0) df_posi3
| PCR 検査陽性者数(単日) | |
|---|---|
| 日付 | |
| 2020-01-16 | 1 |
| 2020-01-17 | 0 |
| 2020-01-18 | 0 |
| 2020-01-19 | 0 |
| 2020-01-20 | 0 |
| ... | ... |
| 2021-05-13 | 6867 |
| 2021-05-14 | 6269 |
| 2021-05-15 | 6420 |
| 2021-05-16 | 5247 |
| 2021-05-17 | 3677 |
488 rows × 1 columns
日付がインデックスになったデータフレームが準備できましたので、次はこの投稿のテーマである期間ごとの集計方法に移ります。
4. resample()メソッドを使った期間の集計
pd.DataFrame.resample()を使うと期間ごとに平均や合計の算出ができる。
第一引数に、
- 週 : W
- 月 : M
- 四半期 : Q
- 年 : Y
などの頻度コードを指定し、更に合計の場合はsum()などのメソッドを呼び出す。
4.1 週ごとに集計
◆週で合計を算出します。その前に違いが分かるように、元ファイルのデータフレームを表示します。
df_posi2
| PCR 検査陽性者数(単日) | |
|---|---|
| 日付 | |
| 2020-01-16 | 1 |
| 2020-01-17 | 0 |
| 2020-01-18 | 0 |
| 2020-01-19 | 0 |
| 2020-01-20 | 0 |
| ... | ... |
| 2021-05-13 | 6867 |
| 2021-05-14 | 6269 |
| 2021-05-15 | 6420 |
| 2021-05-16 | 5247 |
| 2021-05-17 | 3677 |
488 rows × 1 columns
2020-1-16から毎日のデータが488行あります。
◆週で合計を出します。
# 週ごとに合計 df_posi2.resample('W').sum()
| PCR 検査陽性者数(単日) | |
|---|---|
| 日付 | |
| 2020-01-19 | 1 |
| 2020-01-26 | 3 |
| 2020-02-02 | 8 |
| 2020-02-09 | 4 |
| 2020-02-16 | 30 |
| ... | ... |
| 2021-04-25 | 32914 |
| 2021-05-02 | 35836 |
| 2021-05-09 | 35989 |
| 2021-05-16 | 43023 |
| 2021-05-23 | 3677 |
71 rows × 1 columns
2020-01-19から飛び飛びのデータに変わりました。行数も488行から71行に減りました。
どういう日付に変わったのだろうか?
◆日付の曜日を調べてみます。
import datetime # 標準ライブラリ d1 = datetime.date(2020, 1, 19) d2 = datetime.date(2020, 1, 26) d3 = datetime.date(2020, 2, 2) dlst = datetime.date(2021, 5, 23) print(d1.strftime('%A')) print(d2.strftime('%A')) print(d3.strftime('%A')) print(dlst.strftime('%A'))
Sunday
Sunday
Sunday
Sunday
どうやら週ごとに集計すると、全て日曜日の日付で合計が算出されていることが分かりました。
同様に、月、四半期、年でも集計してみます。
4.2 月ごとに集計
# 月別で集計 df_posi2.resample('M').sum()
| PCR 検査陽性者数(単日) | |
|---|---|
| 日付 | |
| 2020-01-31 | 12 |
| 2020-02-29 | 212 |
| 2020-03-31 | 1900 |
| 2020-04-30 | 12361 |
| 2020-05-31 | 2488 |
| 2020-06-30 | 1748 |
| 2020-07-31 | 17367 |
| 2020-08-31 | 32000 |
| 2020-09-30 | 15091 |
| 2020-10-31 | 17583 |
| 2020-11-30 | 47132 |
| 2020-12-31 | 85891 |
| 2021-01-31 | 152585 |
| 2021-02-28 | 41849 |
| 2021-03-31 | 42201 |
| 2021-04-30 | 116476 |
| 2021-05-31 | 94091 |
月の最終日の日付になっております。
4.3 四半期ごとに集計
# 四半期ごとの集計 df_posi2.resample('Q').sum()
| PCR 検査陽性者数(単日) | |
|---|---|
| 日付 | |
| 2020-03-31 | 2124 |
| 2020-06-30 | 16597 |
| 2020-09-30 | 64458 |
| 2020-12-31 | 150606 |
| 2021-03-31 | 236635 |
| 2021-06-30 | 210567 |
四半期は、1月~3月、4月~6月、7月~9月、10月~12月 と1年を3か月ごとで4つに分けますので、日付もそれぞれ3月・6月・9月・12月の最終日となっております。
4.4 年ごとに集計
# 年ごとに集計 df_posi2.resample('Y').sum()
| PCR 検査陽性者数(単日) | |
|---|---|
| 日付 | |
| 2020-12-31 | 233785 |
| 2021-12-31 | 447202 |
日付は大晦日。
4.5 ちなみに、インデックスが日付やdatetimeになっていない場合
結論からいうと、resample(期間コード).sum()を使った方法で集計をする場合、以下の2つの条件だとエラーが出ます。
- インデックスが0から始まる連番がついたデータフレーム
- datetime型に変換しないobject型の日付行をインデックスに変換したデータフレーム
◆2つで集計可能か確認します。(失敗するが( ^ω^)・・・)
# ただ読み込んだだけのデータフレームを年ごと集計 df_ROOT = pd.read_csv(savenames[0]) df_ROOT.resample('Y').sum()
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
<ipython-input-43-cc1c12fb0154> in <module>
1 df_ROOT = pd.read_csv(savenames[0])
2
----> 3 df_ROOT.resample('Y').sum()
~/anaconda3/lib/python3.8/site-packages/pandas/core/generic.py in resample(self, rule, axis, closed, label, convention, kind, loffset, base, on, level, origin, offset)
8075
8076 axis = self._get_axis_number(axis)
-> 8077 return get_resampler(
8078 self,
8079 freq=rule,
~/anaconda3/lib/python3.8/site-packages/pandas/core/resample.py in get_resampler(obj, kind, **kwds)
1267 """
1268 tg = TimeGrouper(**kwds)
-> 1269 return tg._get_resampler(obj, kind=kind)
1270
1271
~/anaconda3/lib/python3.8/site-packages/pandas/core/resample.py in _get_resampler(self, obj, kind)
1432 return TimedeltaIndexResampler(obj, groupby=self, axis=self.axis)
1433
-> 1434 raise TypeError(
1435 "Only valid with DatetimeIndex, "
1436 "TimedeltaIndex or PeriodIndex, "
TypeError: Only valid with DatetimeIndex, TimedeltaIndex or PeriodIndex, but got an instance of 'RangeIndex'
# 日付行をインデックスにしたデータフレームを年ごと集計(省略) #df_ROOT_SET_INDEX = df_ROOT.set_index(df_ROOT['日付']) #df_ROOT_SET_INDEX.resample('Y').sum()
出力はエラーです。省略しました。
TypeError: Only valid with DatetimeIndex, TimedeltaIndex or PeriodIndex, but got an instance of 'Index'
が出ます。
インデックスがdatetime型のデータフレームでなければ期間ごとの集計ができないということが分かりました。
エラーでDatetimeIndexがナンチャラカンチャラと怒られました。
5. 月ごと集計したグラフの作成
最後に、PCR陽性者数とPCR検査実施件数を月ごとに集計し、グラフ化したいと思います。
◆CSVファイルをダウンロードするところからまとめて書きます。
########## ライブラリのインポート ########### import pandas as pd import matplotlib.pyplot as plt ######### データファイルのURL 厚労省/東洋経済より ############ URLs = ["https://www.mhlw.go.jp/content/pcr_positive_daily.csv", # ⓪ PCR陽性者数 厚労省 "https://www.mhlw.go.jp/content/pcr_tested_daily.csv", # ① PCR検査実施人数 厚労省 "https://www.mhlw.go.jp/content/cases_total.csv", # ② 入院治療等を要する者の数 厚労省 "https://www.mhlw.go.jp/content/recovery_total.csv", # ③ 退院又は治療解除となった者の数 厚労省 "https://www.mhlw.go.jp/content/death_total.csv", # ④ 死亡者数合計 厚労省 "https://www.mhlw.go.jp/content/pcr_case_daily.csv", # ⑤ PCR検査の実施件数 厚労省 "https://www.mhlw.go.jp/content/current_situation.csv", # ⑥ 発生状況 厚労省 "https://www.mhlw.go.jp/content/severe_daily.csv", # ⑦ 重傷者数 厚労省 "https://toyokeizai.net/sp/visual/tko/covid19/csv/demography.csv"] # ⑧ 年齢別合計(陽性者、入院者、重症者、死亡者) 東洋経済 ########## ダウンロード読込 ########### # CSVファイル⓪①をダウンロード読込 インデックスをdatetime日付に変更 #df_positive = pd.read_csv(URLs[0], index_col=0, parse_dates=True) # PCR陽性者数データ #df_tested = pd.read_csv(URLs[1], index_col=0, parse_dates=True) # PCR検査実施件数データ ########## ファイル名関数 ########### # CSVファイル名抽出用関数 辞書型 def make_savenames(URLs): savenames = {} for i, fname in enumerate(URLs): savenames[i] = fname.split('/')[-1] return savenames # ローカル保存したファイル名 辞書型 savenames = make_savenames(URLs) ########## ファイルをローカル保存 ########### df_positive.to_csv(savenames[0], index=False) # PCR陽性者数 df_tested.to_csv(savenames[1], index=False) # PCR検査実施件数 ########## ローカルファイル読込(同ファイル名・DatetimeIndexで保存済みの場合のみ使用) ########### #df_positive = pd.read_csv(savenames[0], index_col=0, parse_dates=True) #df_tested = pd.read_csv(savenames[1], index_col=0, parse_dates=True) ########## 月別集計 ########### df_posi_monthly = df_positive.resample('M').sum() # PCR陽性者数 df_tested_monthly = df_tested.resample('M').sum() # PCR検査実施件数 ########## グラフ作図 ########### # DataFrameと画像保存名のリスト df_lst = [df_posi_monthly, df_tested_monthly] jpg_fnames = ["positive", "tested"] # グラフ作図と保存 for d, n in zip(df_lst, jpg_fnames): plt.plot(d) plt.xticks(rotation=30) plt.grid() plt.title(n + "_monthly") plt.savefig(n + "_monthly.jpg") plt.show()
月別陽性者数

月別のPCR検査実施件数
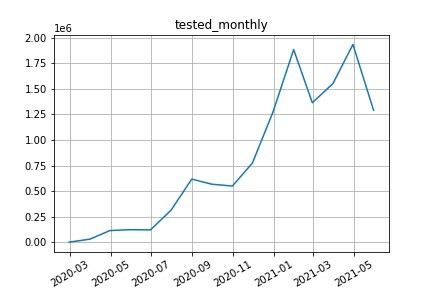
おまけ 陽性率を算出
冬でもないのに2021/05の陽性者数が多い。奇妙です。陽性率を出してみます。
# PCR陽性者数とPCR検査実施件数をマージする _df = pd.merge(df_positive, df_tested, left_index=True, right_index=True, how='outer') _df
| PCR 検査陽性者数(単日) | PCR 検査実施件数(単日) | |
|---|---|---|
| 日付 | ||
| 2020-01-16 | 1 | NaN |
| 2020-01-17 | 0 | NaN |
| 2020-01-18 | 0 | NaN |
| 2020-01-19 | 0 | NaN |
| 2020-01-20 | 0 | NaN |
| ... | ... | ... |
| 2021-05-13 | 6867 | 122502.0 |
| 2021-05-14 | 6269 | 99676.0 |
| 2021-05-15 | 6420 | 66868.0 |
| 2021-05-16 | 5247 | 35948.0 |
| 2021-05-17 | 3677 | 95425.0 |
488 rows × 2 columns
# 欠損値NaNを削除 _df = _df.dropna() # 月別で集計 _df = _df.resample('M').sum() _df # 3列目に陽性率を追加 _df['陽性率'] = _df.iloc[:, 0] / _df.iloc[:, 1] _df
| PCR 検査陽性者数(単日) | PCR 検査実施件数(単日) | 陽性率 | |
|---|---|---|---|
| 日付 | |||
| 2020-02-29 | 210 | 1560.0 | 0.134615 |
| 2020-03-31 | 1796 | 30314.0 | 0.059247 |
| 2020-04-30 | 12137 | 114333.0 | 0.106155 |
| 2020-05-31 | 2488 | 123017.0 | 0.020225 |
| 2020-06-30 | 1748 | 120995.0 | 0.014447 |
| 2020-07-31 | 17367 | 314162.0 | 0.055280 |
| 2020-08-31 | 32000 | 617602.0 | 0.051813 |
| 2020-09-30 | 15091 | 567450.0 | 0.026594 |
| 2020-10-31 | 17583 | 549725.0 | 0.031985 |
| 2020-11-30 | 47132 | 773617.0 | 0.060924 |
| 2020-12-31 | 85891 | 1273942.0 | 0.067421 |
| 2021-01-31 | 152585 | 1883481.0 | 0.081012 |
| 2021-02-28 | 41849 | 1364325.0 | 0.030674 |
| 2021-03-31 | 42201 | 1551196.0 | 0.027205 |
| 2021-04-30 | 116476 | 1933709.0 | 0.060235 |
| 2021-05-31 | 94091 | 1290447.0 | 0.072913 |
# 陽性率グラフ表示 plt.plot(_df.iloc[:,2]) plt.xticks(rotation=30) plt.title('positive_rate') plt.grid() plt.savefig("monthly_positive_rate.jpg") plt.show()
月別の陽性率
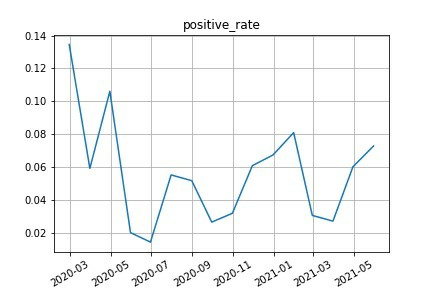
2020年の最初のころは検査数が非常に少ないので陽性率が高いのだが、2021年4月からまた上がってきているのはどういうことなんだろうか。
ワクチンで陽性者が増えてる? ワクチン打つ前に検査するのであれば陽性者数も増えるだろうし、医療関係者は無症状だとしても陽性者は多いだろう。
どういう年齢層やどういう所でどういう人たちを検査しているかが分からないと見えてきませんね。
【参考リンク】
長くなりましたが、以上です。